R语言glmnet包lasso回归中分类变量的处理图文详解
天桥下的卖艺者 人气:0我们在既往文章《手把手教你使用R语言做LASSO 回归》中介绍了glmnet包进行lasso回归,后台不少粉丝发信息向我问到分类变量处理的问题,我后面查了一下资料之前文章分类变量没有处理,非常抱歉。现在来重新聊一聊分类变量的处理。
我们导入glmnet包的时候可以看到,还需要导入一个Matrix包,说明这个矩阵包很重要

按照glmnet包的原文如下:

就是告诉我们,除了Cox Model外,其他的表达都支持矩阵形式,在Cox Model的介绍中,函数样式为


说明我们应该把其他变量变为矩阵的形式。这样说得不是很明白,下面我们来举个例子说明,继续使用我们的乳腺癌数据(公众号回复:乳腺癌,可以获得数据)我们先导入数据和R包
library(glmnet)
library(foreign)
library("survival")
bc <- read.spss("E:/r/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
bc <- na.omit(bc)
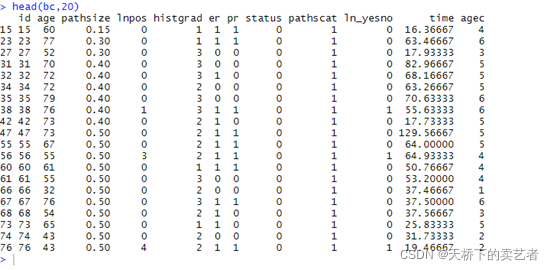
我们先来看看数据:
age表示年龄,pathsize表示病理肿瘤大小(厘米),lnpos表示腋窝淋巴结阳性,histgrad表示病理组织学等级,er表示雌激素受体状态,pr表示孕激素受体状态,status结局事件是否死亡,pathscat表示病理肿瘤大小类别(分组变量),ln_yesno表示是否有淋巴结肿大,time是生存时间,后面的agec是我们自己设定的,不用管它。
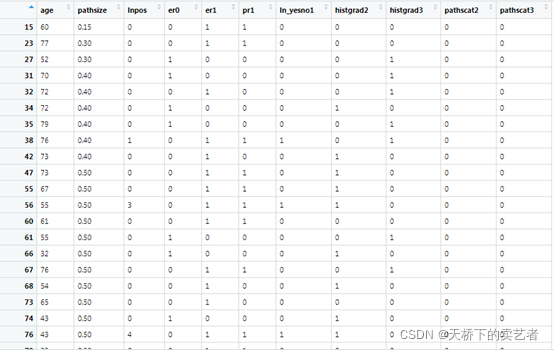
接下来删除缺失变量和把分类变量转成因子
bc$er<-as.factor(bc$er) bc$pr<-as.factor(bc$pr) bc$ln_yesno<-as.factor(bc$ln_yesno) bc$histgrad<-as.factor(bc$histgrad) bc$pathscat<-as.factor(bc$pathscat)
我们先来进行一个lasso的cox模型
glmnet包只能接受矩阵形式的数据,我们要分别进行转换
先把结局和时间提取出来
y<-bc$status time<-bc$time
把id,结局变量,时间变量和一个乱七八糟的变量删掉
data1<-bc[,-c(1,8,11,12)]##把id,结局变量,时间变量和一个乱七八糟的变量删掉
把分类变量变成哑变量矩阵形式
model_mat <-model.matrix(~ +er+pr+ln_yesno+histgrad+pathscat-1,data1)###把分类变量变成哑变量矩阵形式
重新组成数据,也就是我们需要的x
x<-as.matrix(data.frame(age=data1$age,
pathsize=data1$pathsize,lnpos=data1$lnpos,model_mat))#重新组合成数据
弄好x就可以进行分析了,交叉验证最好设一个种子,
set.seed(123) cv.fit <- cv.glmnet(x,Surv(time,y),family="cox", maxit = 1000) plot(cv.fit)

maxit = 1000是让它迭代100次的意思,如果迭代没到1000次,可能会出现一次报错,这在官方说明里面也有讲到,但我用两种方法算了一遍,结果都是一样的,没有错

下图是官方说明
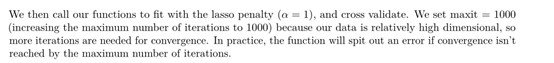
有兴趣的可以试一下这样算,结果也是一样的,但也要先设一个种子
set.seed(123) cv.fit1<- cv.glmnet(x,Surv(time,y),family="cox", alpha=1,nfolds=10) plot(cv.fit1)
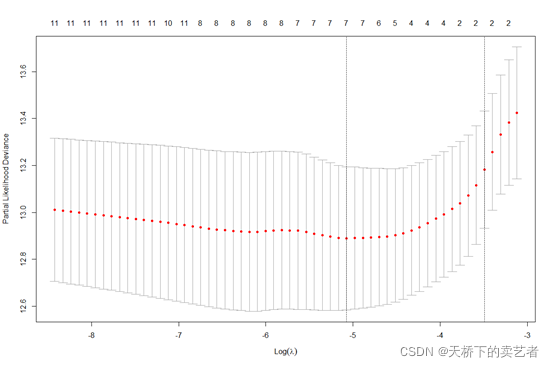
取最小值,也都是一样的
cv.fit$lambda.min cv.fit1$lambda.min

fit <- glmnet(x, Surv(time,y), family = "cox", maxit = 1000) plot(fit)

查看和提取系数
Coefficients <- coef(fit, s = cv.fit$lambda.min) Active.Index <- which(Coefficients != 0) Active.Coefficients <- Coefficients[Active.Index] Active.Index Active.Coefficients
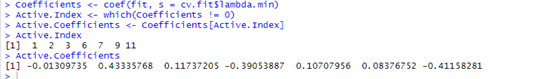
上图标出了最后还剩下的变量(指的是它的位置)和变量的系数,自己对照x看一下就可以了。值得一提的是我看到官方的示例cox模型只取最小的lambda,这样大家就不用这么纠结了,还有一个是它没有预测功能,不能进行预测。
下面来进行Binomial Models,也就是我们的二分类变量模型,其实就是不用时间变量就行了,其他都差不多,继续拿乳腺癌数据演示,懒得找数据了,上一篇文章就是拿乳腺癌来模拟二分类数据的(当时没找到好的数据)。
fit1 = glmnet(x, y, family = "binomial") plot(fit1, xvar = "dev", label = TRUE)

换成lambda
plot(fit1, xvar="lambda", label=TRUE)
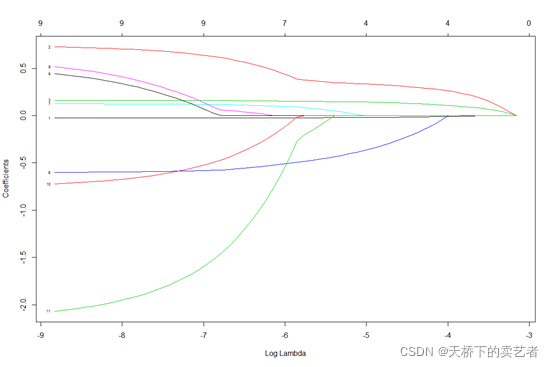
其实到了这里基本和上一篇差不多了
set.seed(999) cvfit=cv.glmnet(x,y, family = "binomial") plot(cvfit)
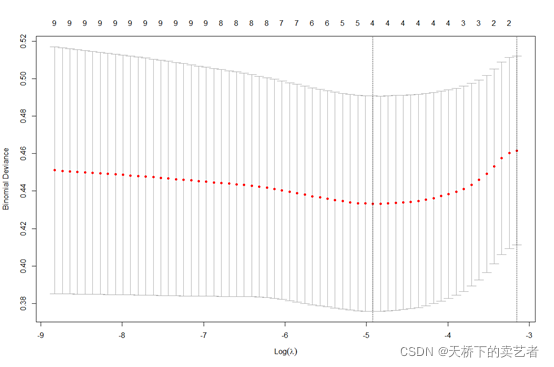
求出最小值
cvfit$lambda.min#求出最小值 cvfit$lambda.1se#求出最小值一个标准误的λ值

求出系数
coef1<-coef(cvfit, s = "lambda.min") coef2<-coef(cvfit, s = "lambda.1se") coef1 coef2

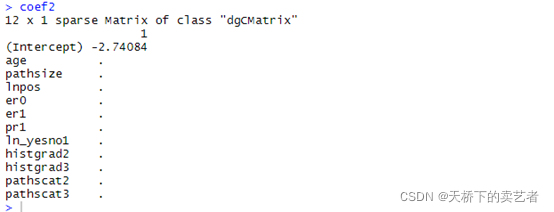
有一个已经被怼没有了,只能选coef1了。
加载全部内容