R语言行筛选filter函数
育种数据分析之放飞自我 人气:0下面介绍一下R语言中行筛选的方法,主要介绍filter函数
1. 数据
这里,使用asreml分析中的BLUP值为例,相关的模型为:
m1 = asreml(Phen ~ G , random = ~ vm(Progeny,ainv) + vm(Dam,ainv) + vm(Progeny,dinv),
workspace = "10Gb", residual = ~ idv(units),data = dat)
summary(m1)$varcomp

计算育种值:
blup = coef(m1)$random head(blup) tail(blup)
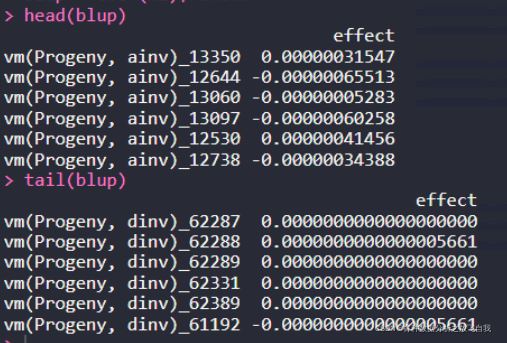
数据特点:
- 没有ID列,rownames的前缀为类型,比如
vm(Progeny, ainv)为加性效应的BLUP值,vm(Progeny,dinv)为显性效应的BLUP值。
提取目的:
- 提取加性效应的BLUP值,显性效应的BLUP值和母体效应的BLUP
- 值提取BLUP值大于0.1的个体
2. 生成ID列和类型
首先,把rowname提取,作为新的一列
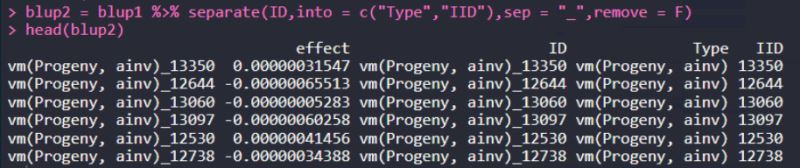
blup1 = blup %>% as.data.frame() %>% mutate(ID = rownames(.)) head(blup1)

根据下划线,进行分列:
blup2 = blup1 %>% separate(ID,into = c("Type","IID"),sep = "_",remove = F)
head(blup2)
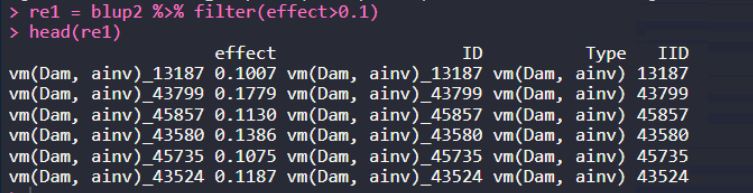
3. 提取effect大于0.1的行
re1 = blup2 %>% filter(effect>0.1) head(re1)
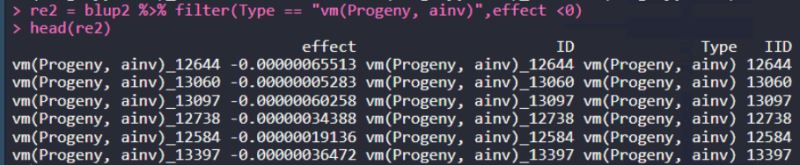
4. 提取加性效应,且effect小于0的行
这里,条件之间,默认是并集,如果想用交集,用|间隔。
re2 = blup2 %>% filter(Type == "vm(Progeny, ainv)",effect <0) head(re2)
5. 根据部分行名删选
select函数,可以根据开头,中间,结尾,进行列的删选。
filter结合其它函数,也可以进行行的筛选。
如果想对ID中,包含ainv的行,进行筛选,可以这样操作:
re3 = blup2 %>% filter(str_detect(ID,"ainv")) %>% arrange(-effect) head(re3)

注意,这里str_detect的pattern是正则表达式。如果直接用原始的字符:
re3 = blup2 %>% filter(str_detect(ID,"vm(Progeny, ainv)")) %>% arrange(-effect) head(re3)
可以看到,报错,如果想要支持,需要对括号用两个反斜线进行转义。
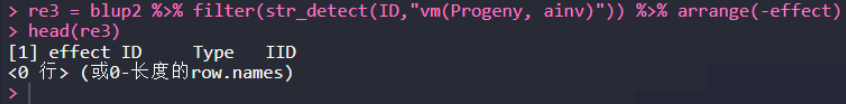
转义后的代码:
re3 = blup2 %>% filter(str_detect(ID,"vm\\(Progeny, ainv\\)")) %>% arrange(-effect) head(re3)

6. 固定字符特征进行行筛选
str_detect没有fixed = T的选项,如果想固定字符匹配,可以用fixed()函数:
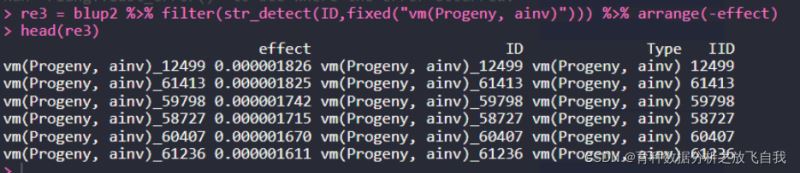
re3 = blup2 %>% filter(str_detect(ID,fixed("vm(Progeny, ainv)"))) %>% arrange(-effect)
head(re3)
总结
加载全部内容